Актуарный анализ выживаемости, разделенный на интервалы
Я пытаюсь создать актуарный анализ выживания в R (я следую некоторым отработанным примерам). Я думаю, что лучший способ сделать это-использовать пакет survival. Итак, что-то вроде:
library(survival)
surv.test <- survfit(Surv(TIME,STATUS), data=test)
TIME на 365 дневных интервалов, и я не могу точно решить, как это сделать, чтобы она соответствовала данному результату.
Насколько я могу понять, в функции survfit нет опции, которая сделает это. Я прошел через несколько примеры документов, и ни один из них не пытался создать лестничный тип участка (есть вариант type='interval', но, кажется, делает что-то другое). Итак, я предполагаю, что мне нужно перегруппировать мои данные, прежде чем я применю функцию survival?
Есть идеи?
P. S: в SPSS это будет INTERVAL = THRU 10000 BY 365; в Stata intervals(365) ... connect(stairsteps)
3 ответа:
Я предполагаю, что вы хотите разделить переменную
TIMEна интервалы, потому что вы хотите построить кривую Каплана-Мейера. В R это не обязательно, вы можете просто вызвать plot на объектеsurvfit. Например,s=survfit(Surv(futime, fustat)~rx, data=ovarian) plot(s)
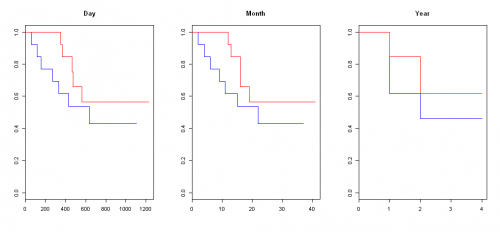
Я думаю, что понимаю ваш вопрос немного лучше. Причина, по которой вы получаете толстую черную линию, заключается в том, что у вас много цензуры, и+строится в каждой отдельной точке, где есть цензура, вы можете отключить это сmark.time=F. (Вы можете увидеть другие варианты в?survival:::plot.survfit) Однако, если вы все еще хотите агрегировать по годам, просто разделите время наблюдения на 365 и округлите.ceilingиспользуется для округления. Вот пример агрегирования на разных временных уровнях без цензуры.Но я думаю, что построение графика Каплана-Мейера без символов цензуры будет выглядеть очень красиво и даст больше понимания.par(mfrow=c(1,3)) plot(survfit(Surv(ceiling(futime), fustat)~rx, data=ovarian),col=c('blue','red'),main='Day',mark.time=F) plot(survfit(Surv(ceiling(futime/30), fustat)~rx, data=ovarian),col=c('blue','red'),main='Month',mark.time=F) plot(survfit(Surv(ceiling(futime/365), fustat)~rx, data=ovarian),col=c('blue','red'),main='Year',mark.time=F) par(mfrow=c(1,1))
Ура, теперь я смогу опубликовать изображения:
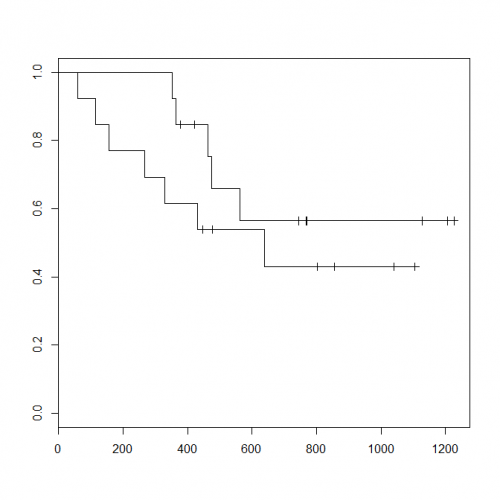
1) Вот как выглядит на данный момент основной сюжет выживания R
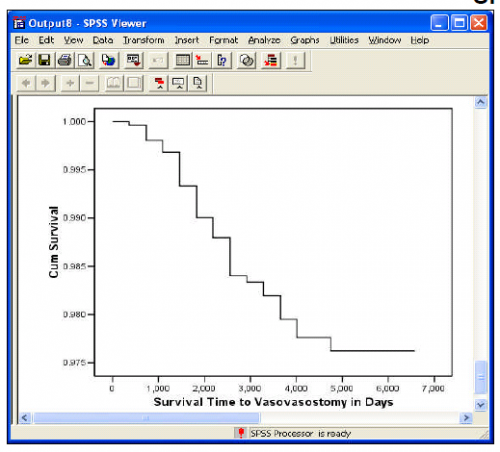
2) и вот как это должно выглядеть (пример SPSS)
Это было именно то, что мне не хватало! Спасибо!
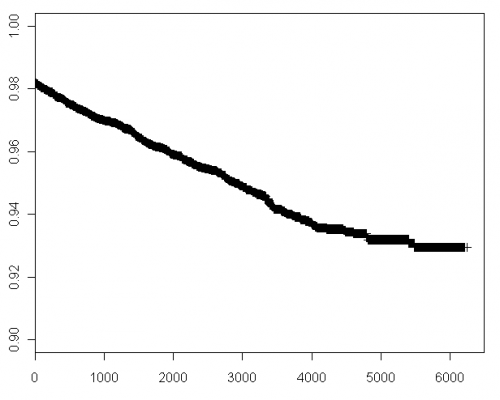
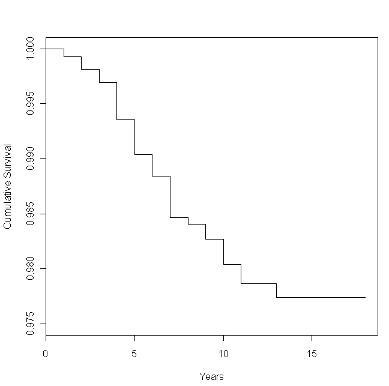
Решение:
vas.surv <- survfit(Surv(ceiling(TIME/365), STATUS)~1, conf.type="none", data=vasectomy) plot(vas.surv, ylim=c(0.975,1), mark.time=F, xlab="Years", ylab="Cumulative Survival")Хорошим штрихом было бы отображение дней на оси x вместо лет (как в Примере SPSS), но я не слишком беспокоюсь об этом.