Плотностный график для нескольких групп в ggplot
Я видел example1 и Как наложить графики плотности в R? и наложенные плотностные графики в ggplot2 о том, как сделать плотностный график. Я могу сделать плотностный график с кодами во втором звене. Однако мне интересно, как я могу сделать такой граф в ggplot или plotly?
Я просмотрел все примеры, но не могу понять, что это за проблема.
У меня есть игрушечный фрейм данных с описанием экспрессии генов лейкоза , столбцы которого в нем относятся к 2 группы лиц
leukemia_big <- read.csv("http://web.stanford.edu/~hastie/CASI_files/DATA/leukemia_big.csv")
df <- data.frame(class= ifelse(grepl("^ALL", colnames(leukemia_big),
fixed = FALSE), "ALL", "AML"), row.names = colnames(leukemia_big))
plot(density(as.matrix(leukemia_big[,df$class=="ALL"])),
lwd=2, col="red")
lines(density(as.matrix(leukemia_big[,df$class=="AML"])),
lwd=2, col="darkgreen")
1 ответ:
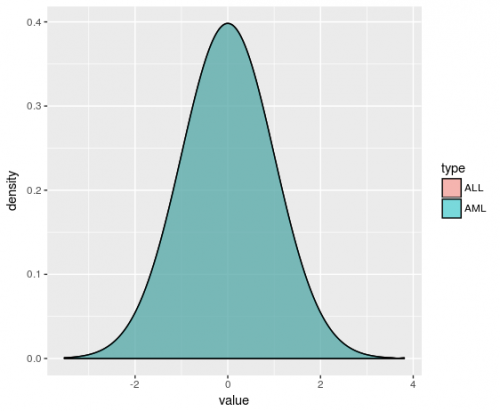
Ggplot требует кругленькую данных, отформатированный, также известный как долгий отформатированные таблицы данных. Это можно сделать в следующем примере. Но будьте осторожны, предоставленный набор данных имеет почти одинаковое распределение значений по типу пациента, таким образом, когда вы строите все типы пациентов и AML, кривые перекрываются, и вы не можете увидеть разницу.
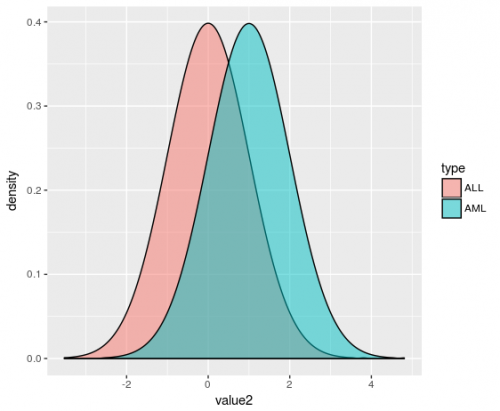
library(tidyverse) leukemia_big %>% as_data_frame() %>% # Optional, makes df a tibble, which makes debugging easier gather(key = patient, value = value, 1:72) %>% #transforms a wide df into a tidy or long df mutate(type = gsub('[.].*$','', patient)) %>% #creates a variable with the type of patient ggplot(aes(x = value, fill = type)) + geom_density(alpha = 0.5)В этом втором примере я добавлю 1 единицу к переменной value для всех пациентов типа AML, чтобы наглядно продемонстрировать проблема перекрытия
leukemia_big %>% as_data_frame() %>% # Optional, makes df a tibble, which makes debugging easier gather(key = patient, value = value, 1:72) %>% #transforms a wide df into a tidy or long df mutate(type = gsub('[.].*$','', patient)) %>% #creates a variable with the type of patient mutate(value2 = if_else(condition = type == "ALL", true = value, false = value + 1)) %>% # Helps demonstrate the overlapping between both type of patients ggplot(aes(x = value2, fill = type)) + geom_density(alpha = 0.5)`