создание нескольких точечных диаграмм с одинаковыми осями в R
Я пытаюсь построить четыре точечных графика в расположении 2 x 2 в R (на самом деле я строю через rpy2). Я бы хотел, чтобы каждый из них имел соотношение сторон 1, но также был на одном и том же масштабе, так что одинаковые X и Y тики для всех вложенных диаграмм, чтобы их можно было сравнить. Я попытался сделать это с помощью par:
par(mfrow=c(2,2))
# scatter 1
plot(x, y, "p", asp=1)
# scatter 2
plot(a, b, "p", asp=1)
# ...
Редактировать:
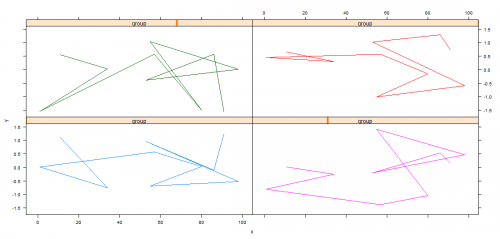
Вот прямой пример того, что я имею сейчас:> par(mfrow=c(2,2))
> for (n in 1:4) { plot(iris$Petal.Width, rnorm(length(iris$Petal.Width)), "p", asp=1) }
Который создает правильный тип рассеяния, но с разными масштабами. Настройка ylim и xlim быть одинаковым в каждом вызове к plot выше не решает проблему. Вы все еще получаете очень разные деления и номера делений на каждой оси, что делает разброс излишне трудным для интерпретации. Я хочу, чтобы оси X и Y были идентичны. Например, это:
for (n in 1:4) { plot(iris$Petal.Width, rnorm(length(iris$Petal.Width)), "p", asp=1, xlim=c(-4, 6), ylim=c(-2, 4)) }
Генерирует неверный результат:
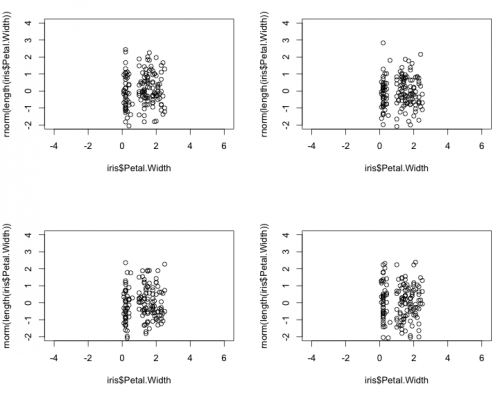
Как лучше всего обеспечить, чтобы одни и те же оси использовались во всех вложенных диаграммах?
Все, что я искал, - это такой параметр, как axis=same или что-то вроде этого для par(mfrow=...), что звучит как поведение по умолчанию для lattice, чтобы сделать оси общими и идентичными в каждом подзаголовке.
plot и явным образом
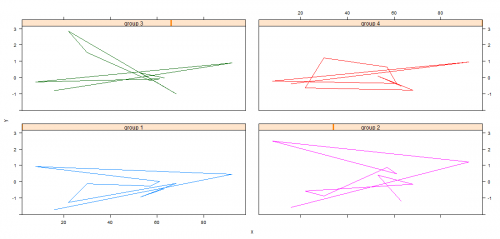
Агстуди дал решение с решеткой. Это выглядит ближе всего к тому, что я хочу, потому что вам не нужно явно предварительно вычислять позиции тиков для каждого разброса, но как новый пользователь я не могу понять, как сделать решетку похожей на обычный график. Самое близкое, что я получил, это:
> xyplot(y~x|group, data =dat, type='p',
between =list(y=2,x=2),
layout=c(2,2), aspect=1,
scales =list(y = list(relation='same'), alternating=FALSE))
Который дает:
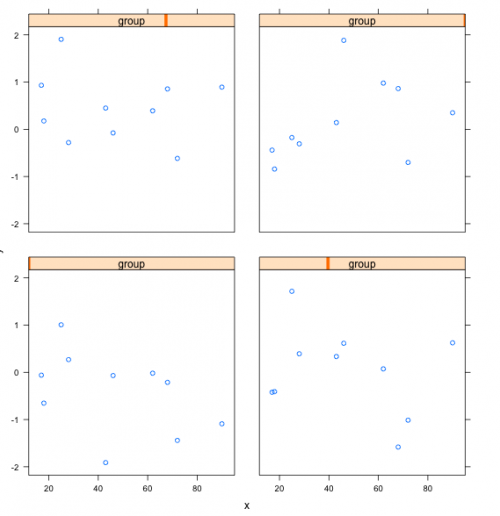
Как я могу сделать это похожим на базу R? Я не хочу, чтобы эти group субтитры были вверху каждого подзаголовка или тики я просто хочу, чтобы каждый x и y скаттера были помечены без маркировки на верхней и правой сторонах каждого скаттера. Я также не ищу общую метку для X и Y - каждый подзаголовок получает свои собственные метки X и Y. И метки оси должны быть одинаковыми в каждом разбросе, хотя с выбранными здесь данными это не имеет смысла.
Если нет простого способа сделать шпалеру похожей на R-базу, то ответ звучит так: нет никакого способа сделать то, что я пытаюсь сделать в R (удивительно), без предварительного вычисления точных мест каждого тика в каждом подзаголовке, что требует предварительного перебора данных.
3 ответа:
Ggplot2 может иметь самое высокое соотношение симпатичный / легкий, если начало.
Пример с rpy2:
from rpy2.robjects.lib import ggplot2 from rpy2.robjects import r, Formula iris = r('iris') p = ggplot2.ggplot(iris) + \ ggplot2.geom_point(ggplot2.aes_string(x="Sepal.Length", y="Sepal.Width")) + \ ggplot2.facet_wrap(Formula('~ Species'), ncol=2, nrow = 2) + \ ggplot2.GBaseObject(r('ggplot2::coord_fixed')()) # aspect ratio # coord_fixed() missing from the interface, # therefore the hack. This should be fixed in rpy2-2.3.3 p.plot()Читая комментарии к предыдущему ответу, я вижу, что вы можете иметь в виду совершенно отдельный график. С системой построения графиков по умолчанию для R,
par(mfrow(c(2,2))илиpar(mfcol(c(2,2)))будет проще всего пойти и сохранить соотношение сторон, диапазоны для осей и делений последовательными через обычный способ, которым они фиксируются.Наиболее гибкой системой для построения графика в R может быть
grid. Это не так плохо, как кажется. кажется, подумайте о том, как выглядит график сцены. С помощью rpy2, ggplot2 и сетки:from rpy2.robjects.vectors import FloatVector from rpy2.robjects.lib import grid grid.newpage() lt = grid.layout(2,2) # 2x2 layout vp = grid.viewport(layout = lt) vp.push() # limits for axes and tickmarks have to be known or computed beforehand xlims = FloatVector((4, 9)) xbreaks = FloatVector((4,6,8)) ylims = FloatVector((-3, 3)) ybreaks = FloatVector((-2, 0, 2)) # first panel vp_p = grid.viewport(**{'layout.pos.col':1, 'layout.pos.row': 1}) p = ggplot2.ggplot(iris) + \ ggplot2.geom_point(ggplot2.aes_string(x="Sepal.Length", y="rnorm(nrow(iris))")) + \ ggplot2.GBaseObject(r('ggplot2::coord_fixed')()) + \ ggplot2.scale_x_continuous(limits = xlims, breaks = xbreaks) + \ ggplot2.scale_y_continuous(limits = ylims, breaks = ybreaks) p.plot(vp = vp_p) # third panel vp_p = grid.viewport(**{'layout.pos.col':2, 'layout.pos.row': 2}) p = ggplot2.ggplot(iris) + \ ggplot2.geom_point(ggplot2.aes_string(x="Sepal.Length", y="rnorm(nrow(iris))")) + \ ggplot2.GBaseObject(r('ggplot2::coord_fixed')()) + \ ggplot2.scale_x_continuous(limits = xlims, breaks = xbreaks) + \ ggplot2.scale_y_continuous(limits = ylims, breaks = ybreaks) p.plot(vp = vp_p)Больше документации в документацииrpy2 о графике , а затем в документации ggplot2 и grid.
С помощью
latticeиggplot2необходимо изменить форму данных. Например:
- создайте 4 данных.рамка (x=x1, y=y1)...
- добавьте столбец группы для каждого типа данных.рамка, группа=1,2,...
- rbind данные 4.кадр в один раз
Вот пример использования
latticedat <- data.frame(x = rep(sample(1:100,size=10),4), y = rep(rnorm(40)), group = rep(1:4,each =10)) xyplot(y~x|group, ## conditional formula to get 4 panels data =dat, ## data type='l', ## line type for plot groups=group, ## group ti get differents colors layout=c(2,2)) ## equivalent to par or layoutPS: нет необходимости устанавливать сакли. В
xyplotнастройки sacles по умолчанию -same(одинаковые sacles для всех панелей). Вы можете изменить его, например :xyplot(y~x|group, data =dat, type='l',groups=group, layout=c(2,2), scales =list(y = list(relation='free')))EDIT
Существует большое количество аргументов для построения решетки функции, позволяющие контролировать многие детали сюжета, вот например я настраиваю:
- Текст, используемый для надписей и заголовков для полос
- размер и размещение меток галочек оси,
Размер зазоров между столбцами и рядами панелей.
xyplot(y~x|group, data =dat, type='l',groups=group, between =list(y=2,x=2), layout=c(2,2), strip = myStrip, scales =list(y = list(relation='same',alternating= c(3,3))))Где
myStrip <- function(var.name,which.panel, which.given,...) { var.name <- paste(var.name ,which.panel) strip.default(which.given,which.panel,var.name,...) }
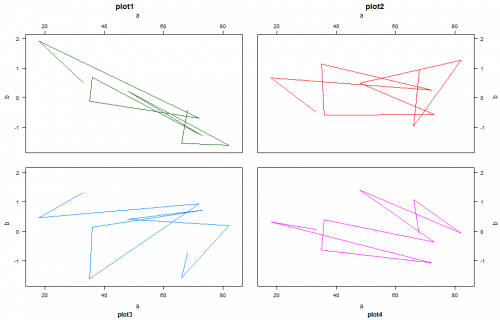
EDIT In для того чтобы получить решетку сюжета base-graphics plots, вы можете попробовать следующее:
xyplot(y~x|group, data =dat, type='l',groups=group, between=list(y=2,x=2), layout=c(2,2), strip =FALSE, xlab=c('a','a'), xlab.top=c('a','a'), ylab=c('b','b'), ylab.right = c('b','b'), main=c('plot1','plot2'), sub=c('plot3','plot4'), scales =list(y = list(alternating= c(3,3)), x = list(alternating= c(3,3))))
Хотя ответ уже выбран, этот ответ использует
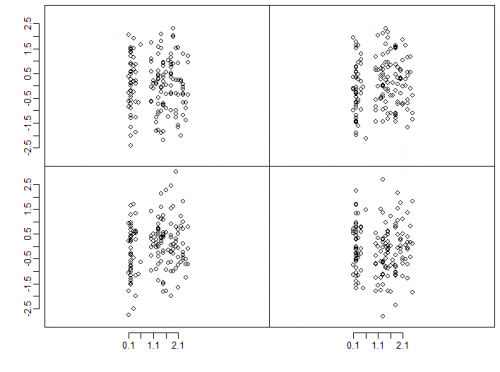
Я бы предложил прочитать объяснение Шона Андерсона магии, которую можно использовать с умным использованиемggplot, а не базу R, что и требовалось OP. Хотяggplotдействительно хорош для быстрого построения графиков, для публикации часто требуется более тонкий контроль над сюжетами, чем предлагаетggplot. Вот где базовый сюжет выделяется.par, а также несколько других хороших трюков, таких как использованиеlayout()иsplit.screen(). Используя его объяснение, я пришел к следующему выводу:# Assume that you are starting with some data, # rather than generating it on the fly data_mat <- matrix(rnorm(600), nrow=4, ncol=150) x_val <- iris$Petal.Width Ylim <- c(-3, 3) Xlim <- c(0, 2.5) # You'll need to make the ylimits the same if you want to share axes par(mfrow=c(2,2)) par(mar=c(0,0,0,0), oma=c(4,4,0.5,0.5)) par(mgp=c(1, 0.6, 0.5)) for (n in 1:4) { plot(x_val, data_mat[n,], "p", asp=1, axes=FALSE, ylim=Ylim, xlim=Xlim) box() if(n %in% c(1,3)){ axis(2, at=seq(Ylim[1]+0.5, Ylim[2]-0.5, by=0.5)) } if(n %in% c(3,4)){ axis(1, at=seq(min(x_val), max(x_val), by=0.1)) } }
Здесь еще есть над чем поработать. Так же, как и в ОП, данные выглядят раздавленными посередине. Конечно, было бы неплохо настроить все так, чтобы использовалась вся область построения.