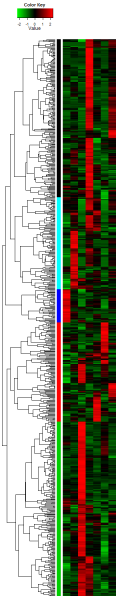
Как развернуть дендограмму в heatmap.Два
У меня есть следующая тепловая карта с такой дендрограммой.
Полные данные находятсяздесь .
Проблема в том, что дендрограмма слева раздавлена. Как я могу отменить (развернуть) его, не изменяя размер столбца тепловой карты?
Он генерируется с помощью следующего кода:
#!/usr/bin/Rscript
library(gplots);
library(RColorBrewer);
plot_hclust <- function(inputfile,clust.height,type.order=c(),row.margins=70) {
# Read data
dat.bcd <- read.table(inputfile,na.strings=NA, sep="t",header=TRUE);
rownames(dat.bcd) <- do.call(paste,c(dat.bcd[c("Probes","Gene.symbol")],sep=" "))
dat.bcd <- dat.bcd[,!names(dat.bcd) %in% c("Probes","Gene.symbol")]
dat.bcd <- dat.bcd
# Clustering and distance function
hclustfunc <- function(x) hclust(x, method="complete")
distfunc <- function(x) dist(x,method="maximum")
# Select based on FC, as long as any of them >= anylim
anylim <- 2.0
dat.bcd <- dat.bcd[ apply(dat.bcd, 1,function(x) any (x >= anylim)), ]
# Clustering functions
height <- clust.height;
# Define output file name
heatout <- paste("tmp.pafc.heat.",anylim,".h",height,".pdf",sep="");
# Compute distance and clusteirn function
d.bcd <- distfunc(dat.bcd)
fit.bcd <- hclustfunc(d.bcd)
# Cluster by height
#cutree and rect.huclust has to be used in tandem
clusters <- cutree(fit.bcd, h=height)
nofclust.height <- length(unique(as.vector(clusters)));
myorder <- colnames(dat.bcd);
if (length(type.order)>0) {
myorder <- type.order
}
# Define colors
#hmcols <- rev(brewer.pal(11,"Spectral"));
hmcols <- rev(redgreen(2750));
selcol <- colorRampPalette(brewer.pal(12,"Set3"))
selcol2 <- colorRampPalette(brewer.pal(9,"Set1"))
sdcol= selcol(5);
clustcol.height = selcol2(nofclust.height);
# Plot heatmap
pdf(file=heatout,width=20,height=50); # for FC.lim >=2
heatmap.2(as.matrix(dat.bcd[,myorder]),Colv=FALSE,density.info="none",lhei=c(0.1,4),dendrogram="row",scale="row",RowSideColors=clustcol.height[clusters],col=hmcols,trace="none", margin=c(30,row.margins), hclust=hclustfunc,distfun=distfunc,lwid=c(1.5,2.0),keysize=0.3);
dev.off();
}
#--------------------------------------------------
# ENd of functions
#--------------------------------------------------
plot_hclust("http://pastebin.com/raw.php?i=ZaGkPTGm",clust.height=3,row.margins=70);
3 ответа:
В вашем случае данные имеют длинный хвост, который ожидается для данных экспрессии генов (логнормальных).
Как видно из квантильного распределения, гены с наибольшей экспрессией расширяют диапазон от 1,5 до более 300.data <- read.table(file='http://pastebin.com/raw.php?i=ZaGkPTGm', header=TRUE, row.names=1) mat <- as.matrix(data[,-1]) # -1 removes the first column containing gene symbolsquantile(mat) # 0% 25% 50% 75% 100% # 0.000 0.769 1.079 1.544 346.230Когда иерархическая кластеризация выполняется на нескаленных данных, результирующая дендрограмма может показать смещение в сторону значений с наибольшим выражением, как показано в вашем примере. Это заслуживает либо логарифмического, либо преобразование z-score, среди многих (Ссылка). Ваш набор данных содержит
values == 0, что является проблемой для преобразования журнала, так какlog(0)не определено.Преобразование Z-баллов (ссылка ) реализована в
heatmap.2, но важно отметить, что функция вычисляет матрицу расстояний и запускает алгоритм кластеризации перед масштабированием данных. Следовательно, опцияscale='row'не влияет на результаты кластеризации, см. мой предыдущий пост (различия в heatmap / clustering defaults in R ) для получения более подробной информации.Я бы предложил вам масштабировать ваши данные Перед запуском
heatmap.2:# scale function transforms columns by default hence the need for transposition. z <- t(scale(t(mat))) quantile(z) # 0% 25% 50% 75% 100% # -2.1843994 -0.6646909 -0.2239677 0.3440102 2.2640027 # set custom distance and clustering functions hclustfunc <- function(x) hclust(x, method="complete") distfunc <- function(x) dist(x,method="maximum") # obtain the clusters fit <- hclustfunc(distfunc(z)) clusters <- cutree(fit, 5) # require(gplots) pdf(file='heatmap.pdf', height=50, width=10) heatmap.2(z, trace='none', dendrogram='row', Colv=F, scale='none', hclust=hclustfunc, distfun=distfunc, col=greenred(256), symbreak=T, margins=c(10,20), keysize=0.5, labRow=data$Gene.symbol, lwid=c(1,0.05,1), lhei=c(0.03,1), lmat=rbind(c(5,0,4),c(3,1,2)), RowSideColors=as.character(clusters)) dev.off()Также смотрите дополнительные сообщения здесь и здесь , которые объясняют, как установить макет тепловой карты через
Полученная тепловая карта показана ниже (метки строк и столбцов опущены):lmat,lwidиlheiпараметры.
Насколько я могу судить, у вас могут быть некоторые выбросы в вашем наборе данных (объекты в самом низу). Попробуйте следующее:
- Удаление выбросов из набора данных
- логарифмический масштаб вашего расстояния, чтобы дать меньше внимания экстремальным значениям
Преобразование Zscore легко с помощью пакета 'fheatmap', используя параметр 'scale'. Проверьте пакет "fheatmap". Высота dendograms может быть расширена за счет увеличения с холста(в формате PDF). http://cran.r-project.org/web/packages/fheatmap/index.html